밀폐이용 안전관리 기술정보
왼쪽메뉴
밀폐이용 안전관리 기술정보
본문컨텐츠
정량검출법
PMA-qPCR 유전자변형 미생물 정량 검출법
1. 실험목적
- 본 ‘PMA-qPCR 이용한 유전자변형 미생물 정량 검출법’ 실험은 액체 배양된(액체 상태의) 유전자변형(Genetically Modified) 미생물 중 살아있는 유전자변형 미생물(Living Modified Microorganisms, LMMs)을 정량 검출하기 위한 방법이다.
- 본 실험법에서는 미생물로부터 DNA를 추출하지 않고, 균주를 직접 사용하는 direct PCR법과 정량 분석을 위한 TaqMan probe 기반의 dual-plex qPCR 분석, 살아있는 균주만을 검출할 수 있는 PMA (propidium monoazide) 처리 방법이 사용된다.
- 본 실험은 항생제 ampicillin, kanamycin 또는 chloramphenicol 내성 유전자를 포함한 대장균(Escherichia coli)과 코리네박테리움 글루타미쿰(Corynebacterium glutamicum)을 검출하기 위한 목적으로 수행되었다.
- 참고사항
- PMA (propidium monoazide)는 DNA와 결합하여 PCR 증폭을 방해하는 감광성 염료로서, 살아있는 세포막은 투과할 수 없으나 죽은 세포의 손상된 세포막을 투과할 수 있다. 미생물 용액에 PMA를 처리하면 PMA가 죽은 세포 내의 DNA 또는 용액에 나출된 DNA와 결합하여 이들의 위양성 PCR 반응을 방지하여, 결과적으로 살아있는 미생물 세포의 선택적 검출이 가능하도록 한다(Rudi et al., 2005).
2. 실험장비(도구) 및 재료
2.1 실험기자재
- - 냉장고
- - 미생물 항온 배양기
- - 진탕 배양기
- - 볼텍스믹서
- - 실시간 PCR 기기 (StepOne TM real-time PCR system)
- - Micro Pipette (1-10㎕, 20-200㎕, 100㎕-1000㎕)
- - 소형 전기영동장치(Mini Submarine Electrophoresis System)
- - 다용도 원심분리기
- - 얼음 수조 (Ice box), 얼음 PCR 튜브 rack
- - PMA-Lite™LED photolysis device
- - 무균작업대
- - 흡광도계 NanoDrop spectrophotometer
- - PCR tube (0.2㎖)용 Micro centrifuge
2.2 실험재료
- - PCR tube (200㎕)
- - PCR tip (1-10㎕, 20-200㎕, 100㎕-1000㎕)
- - Luria-Berani (LB) broth (E.coli)
- - Luria-Berani (LB), Brain Heart Infusion (BHI) broth (C.glutamicum)
- - 항생제 (50ug/㎖ kanamycin, 50ug/㎖ ampicillin, 25ug/㎖ chloramphenicol)
- - 1.5㎖ 튜브, 50㎖ conical 튜브
- - Nuclease-Free water (멸균증류수)
- - Oligo primer, TaqMan probe
- - 1.5% agarose gel
- - SafePinky DNA gel staining solution (10000×) in water
- - PMA solution (20mM, PMAxxTM Dye)
- - TOPreal™ qPCR 2X PreMIX (TaqMan Probe for multiplex)
- - Wizard® Plus SV Minipreps DNA purification system
- - Wizard® Genomic DNA purification kit
- - 100 bp DNA Ladder
3. 실험과정
3.1 Oligo primer 및 probe 제작
3.1.1 유전자변형 미생물 특이적(event-specific) oligo primer 및 probe 제작
- 1) 유전자변형 미생물을 정확히 식별하기 위해 도입된 유전자와 주변 염기서열을 기반으로 해당 LMM에 특이적인 oligo primer과 probe를 설계하는 것이 권장된다. OligoArchitect TM Online 등의 프로그램을 사용하여 적절한 primer과 probe를 설계할 수 있다.
- 2) qPCR 분석의 효율성을 높이기 위해 PCR product가 90~200 bp의 크기가 되도록 primer을 설계한다. Primer과 probe의 길이는 20~30bp 크기로 조정하고, annealing 온도는 62~68 ℃로 설정한다.
- 3) 항생제 내성 유전자인 ampicillin (AmpR), kanamycin (KmR), chloramphenicol (CmR)을 함유하는 미생물을 검출하기 위한 primer와 probe 정보는 표 1과 같다.
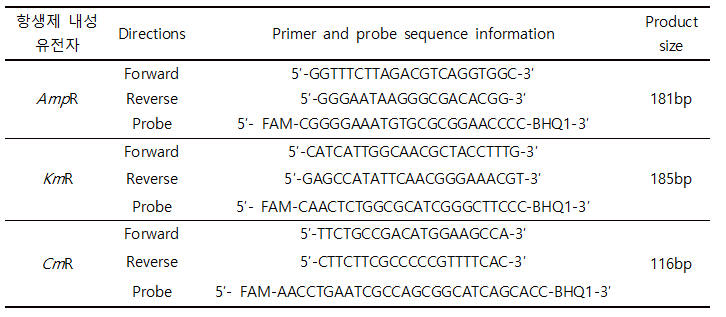
- 표 1. 항생제 내성 유전자에 대한 primer 및 probe 정보 (예시)
3.1.2 유전자변형 미생물 숙주 내재유전자에 대한 oligo primer 및 probe 제작
- 1) 유전자변형 미생물 중 plasmid 형태로 유전자를 도입한 경우 다중 복사본(multi-copy)을 가지고 있는 경우가 많기 때문에 정량 분석 시 미생물의 염색체에 있는 단일 복사본(single copy)의 내재유전자를 기반으로 qPCR의 internal control(분류균 특이적, taxon-specific)을 사용하는 것이 권장된다.
- 2) E. coli의 경우 D-1- deoxyxylulose 5-phosphate synthase gene (dxs, AF035440)이 대표적인 내재유전자로 알려져 있으며, chromosomal replication initiator protein dnaA 유전자는 코리네박테리움 글루타미쿰(C. glutamicum)의 단일 복사본인 내재유전자로 알려져 있다(Lee et al., 2006; Okibe et al., 2010). 이들에 대한 taxon-specific oligo primer과 probe 정보는 표 2와 같다.
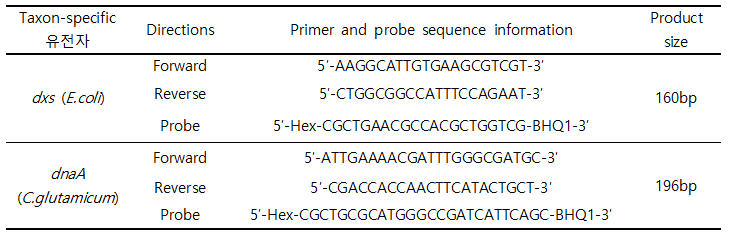
- 표 2. 대장균 (E. coli) 및 코리네박테리움 글루타미쿰(C. glutamicum) 내재유전자 (taxon-specific)에 대한 primer 및 probe 정보 (예시)
3.2 Plasmid 및 genomic DNA추출
3.2.1 DNA 추출
- 1) Plasmid DNA의 경우 Wizard® Plus SV Minipreps DNA purification system 등의 plasmid DNA 추출 키트를 이용하여 각 키트별 표준 프로토콜에 따라 plasmid DNA를 추출한다.
- 2) Genomic DNA의 경우 Wizard®Genomic DNA purification kit 등 해당 미생물에 적합한 genomic DNA 추출 키트를 사용하여 각 키트별 표준 프로토콜에 따라 DNA를 추출한다.
3.2.2 DNA 정량
- 1) Nanodrop spectrophotometer 등을 이용하여 plasmid DNA 및 genomic DNA의 농도를 측정한다. 이때 A260/A280과 A260/A230의 DNA 순도가 1.8 이상이 되어야 한다.
3.3 표준 정량 곡선 (Standard curve) 생산
3.3.1 Standard DNA 준비
- 1) NCBI (National Center for Biotechnology Information) 자료를 검색하여 해당 유전자변형 미생물 숙주의 genomic DNA 크기를 확인한다. 참고로 E. coli 및 C. glutamicum의 genomic DNA 크기는 각각 4,534,037 bp와 3,282,708 bp이다(Song et al., 2015; Kalinowski et al., 2003). Plasmid DNA 크기는 사용된 도입 벡터의 크기에 따라 다를 수 있다.
- 2) Standard DNA 농도(ng/㎕)를 아래의 공식에 따라 ㎕당 copy 수로 전환한다.
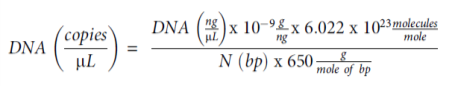
- N (bp), plasmid DNA 또는 genome DNA size; 6.022×1023 molecules/mol, Avogadro’number; 염기 하나의 DNA molecular weight, 650 Da (Whelan et al., 2003).
- 3) 108 copies/㎕ 농도의 plasmid DNA 또는 genomic DNA 현탁액을 제작한다.
- 예시
- Plasmid DNA 크기가 7,683 bp로 알려져 있는 108 copies의 벡터 pJ281 (KmR)의 경우 위의 계산식에 따라 DNA 농도는 0.842 ng이다(108 × 7,683(bp) × 1.096 × 10-21(g)).
- 만약 추출된 DNA 농도가 40ng/㎕일 경우, 108 copies/㎕ 농도의 DNA 용액 100㎕를 만들 때의 계산식은 0.842ng × 100㎕ ÷ 40ng = 2.1㎕이다.
- 따라서 2.1㎕의 DNA 현탁액(40ng/㎕ 농도)에 97.9㎕의 nuclease-free water를 첨가하면 108 copies DNA 샘플을 만들 수 있다.
- 4) 순차적으로 희석된 DNA 용액을 준비한다. Nuclease-free water 이용하여 DNA 농도(copies/㎕)가 107 에서 102가 되도록 한다. DNA 용액의 희석은 30초 동안 vortex 한 108 copies/㎕ 농도의 DNA 현탁액 10㎕를 90㎕의 nuclease-free water에 넣어 107 copies/㎕ 농도의 DNA 샘플을 제작한 후 이후 동일한 방식으로 106, 105, 104, 103, 102 copies/㎕ 농도의 DNA 샘플을 준비한다.
3.3.2 qPCR 수행 및 standard curve(표준 정량 곡선) 생산
- 1) Event-specific과 taxon-specific 유전자에 대해 각각의 primer/probe를 사용하여 standard plasmid DNA와 genomic DNA를 대상으로 single-plex qPCR을 각각 수행한다.
- 2) Single-plex qPCR의 PCR 반응액 조성은 표3과 같으며, 결과값은 그림 1과 같다.

- 표 3. Single-plex qPCR을 위한 PCR 반응용액의 조성표(예시)
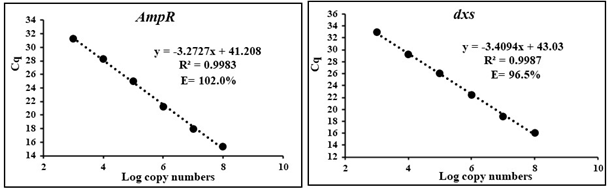
- 그림 1. 유전자변형 대장균의 plasmid DNA 및 genomic DNA의 연속 희석액을 사용하여 qPCR 분석을 통해 AmpR 및 dxs의 표준 정량 곡선 예시(Qin et al., 2022).
- 4) qPCR 분석은 real-time PCR 기기를 이용하여 진행한다. qPCR 반응 조건은 pre-denaturation 95℃ 10분, denaturation 95℃ 30초, annealing 60℃ 30초로 하여 총 40 cycle을 수행한다. qPCR은 3반복으로 2회 수행하며 qPCR RT 값은 평균 Cq값으로 계산한다. Cq값은 시스템의 기본 임계값을 사용하여 자동으로 계산된다.
- 5) NTC (non-template control)는 DNA 현탁액 대신에 nuclease-free water를 첨가한 음성 대조구 시료이며 실제 측정하려는 DNA 시료와 동일한 PCR 조건으로 동시에 qPCR을 수행한다. NTC의 RT 값이 35 이상일 경우 primer/probe의 오염 및 교차반응이 없는 것으로 판단되므로, RT 값이 NTC의 RT 값보다 작은 DNA 시료를 이용하여 유전자변형 미생물의 도입유전자와 내재유전자에 대한 선형 회귀분석을 Excel 프로그램 등을 이용하여 수행한다(그림 1).
3.3.3 Standard curve 유효성 검사
- 1) 생산된 선형 회귀 방정식의 Yinter와 slope를 이용하여 qPCR efficiency (E)를 E = 10 (-1/slope)-1로 계산한다. 유효한 standard curve는 slope의 범위 –3.1 ~ -3.6, R2 >0.95, PCR efficiency E는 90%~110% 사이의 범위 값을 나타내야 한다(European Network of GMO Laboratories (ENGLs), 2015).
- 2) 선형 회귀 방정식은 아래의 공식을 이용하여 희석된 DNA 각각에 대한 DNA copy 수를 계산한다.
- 3) qPCR을 분석할 때 사용된 DNA copy 수와 비교한 bias(%) 값을 구한다. 이때 bias(%) 값은 ±25% 이내여야 한다(표 4). Bias(%) 는 아래 공식으로 계산한다.
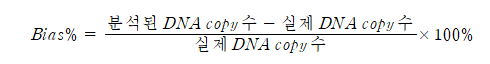
- 4) PCR 반응의 재현성은 모든 희석배수의 DNA 시료가 ±25% 이내의 표준편차(RSD) 값을 나타내는지 확인할 수 있도록 희석 점 반복 간 상대적 RSD(%)를 구한다. 표준 곡선은 E 값, RSD(%) 및 bias(%) 값이 모두 ENGL 에서 지정한 범위 내에 있어야만 사용할 수 있다(그림 1, 표 4).
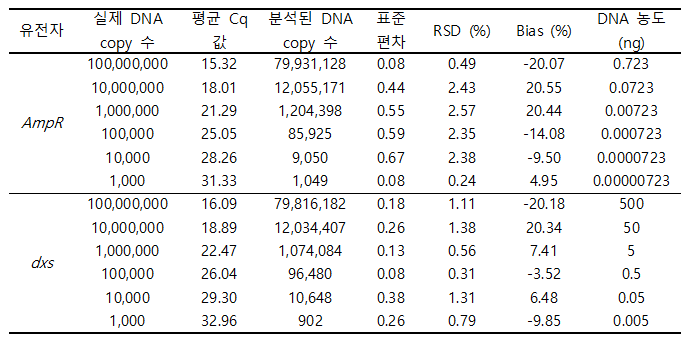
- 표4. 유전자변형 대장균의 plasmid DNA 및 genomic DNA를 이용한 AmpR 및 dxs의 표준 정량 곡선 생산 및 유효성 분석 예시(Qin et al., 2022).
3.4 유전자변형 미생물 배양 및 PMA 처리
3.4.1 미생물 배양
- 1) 유전자변형 미생물을 10㎖ 배양액에 해당 항생제가 첨가된 액체배지에 배양하고 적절한 배양온도에서 진탕배양 한다.
- 2) 배양된 미생물은 분광광도계를 이용하여 600nm에서 광학밀도(OD) 값을 3 반북 측정하고 평균 OD 값이 0.7 이상인 배양액을 다음 단계 실험에 사용한다. 배양액은 얼음에 보관한다.
3.4.2 PMA 처리
- 1) PMA 처리를 위하여 배양액을 희석한다. 잘 혼합된 배양액 100㎕를 1.5㎖ 튜브에 옮겨준 후 900㎕ nuclease-free water를 첨가하여 10-1 희석액을 만든다. 10-1 희석액을 30초간 vortex 한 후 900㎕ nuclease-free water에 100㎕ 10-1 희석액을 넣어 10-2 희석액을 만든다.
- 2) 450㎕의 10-2 세포 희석액을 1.5㎖ 튜브에 옮긴 후 1㎕의 PMA solution (20mM)을 넣고 10초간 vortex 한 후 암실에서 5분간 180rpm의 속도로 진탕 배양한다.
- 3) PMA-Lite™ LED Photolysis Device를 이용하여 15분간 청색광 처리한다.
- 4) PMA 처리된 샘플은 얼음에 차광 보관한다.
3.5 PMA - qPCR 분석
3.5.1 Cell-direct dual-plex qPCR 분석
- 1) Cell-direct qPCR은 PMA 처리된 세포 현탁액을 DNA template로 사용한다. 각 시료는 30초 동안 vortex 한 후에 다음 단계를 진행한다.
- 2) Dual-plex PMA-qPCR 반응액 조성은 표5과 같다.
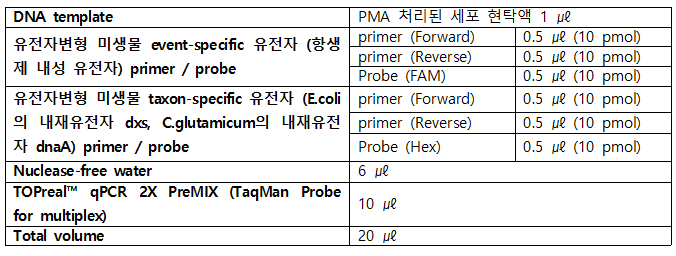
- 표 5 Dual-plex PMA-qPCR을 위한 PCR 반응용액의 조성표 (예시)
- 3) PMA-qPCR 분석은 real-time PCR기를 이용한다. PMA-qPCR 반응 조건은 pre-denaturation 95℃ 10분, denaturation 95℃ 30초, annealing 60℃ 30초로 하여 총 40 cycle을 수행한다.
- 4) PMA-qPCR은 3 반복으로 수행하며 각 시료의 qPCR RT 값은 평균 Cq 값으로 계산한다. Cq 값은 시스템의 기본 임계값을 사용하여 자동으로 계산된다 (이하 Cq 값은 PMA-Cq로 표시한다).
- 5) NTC(non-template control)는 nuclease-free water를 첨가한 음성 대조구(컨트롤) 시료이며 실제 측정하려는 PMA 처리된 세포 현탁액과 동일한 PCR 조건으로 동시에 qPCR을 수행한다. NTC의 Cq 값이 모두35 이상이어야 qPCR 분석이 오염되지 않는 것으로 판단된다.
3.5.2 살아있는 유전자변형 미생물 정량 분석
- 1) 각 시료의 event-specific 유전자의 평균 PMA-Cq값을 이용해서 event-specific 유전자의 copy 수를 계산하면 해당 유전자변형 미생물 현탁액에서 살아있는 미생물의 plasmid copy 수를 구할 수 있다. 또한 각 시료의 taxon-specific 유전자의 평균 PMA-Cq값을 이용해서 taxon-specific 유전자의 copy 수를 계산하면 해당 유전자변형 미생물에 살아있는 균주 세포 수를 구할 수 있다.
- 2) 각 평균 PMA-Cq값을 이용하여 앞서 3.3.3에서 standard plasmid DNA과 genomic DNA로 생산된 event-specific과 taxon-specific 유전자에 대한 표준정량곡선을 사용하여 아래 공식을 따라 해당 유전자변형 미생물에 살아있는 세포의 plasmid copy 수와 균주 세포 수를 계산한다.
- DNA copies = 10 (PMA-Cq value – Yinter ) / slope × 100 (여기서 100은 PMA 처리된 배양액의 희석배수임)
- 예시
- Ampicillin 항생제 내성 유전자변형 대장균의 배양액을 이용해서 10-2의 희석액을 제작하였다. PMA 처리 후 Ampicillin 내성 유전자 AmpR 과 대장균 내재유전자 dxs에 대한 dual-plex qPCR 분석을 수행하였다.
- AmpR 유전자의 경우, 10-2 희석액의 qPCR 평균 PMA-Cq값은 23.24로 계산되었다. 앞서 그림1에서 AmpR의 표준정량 곡선은 Y = -3.2727x + 41.208로 생산되었다. 이 경우 유전자변형 대장균에 살아있는 세포의 plasmid copy 수는 10(23.24-41.208)÷-3.2727 × 100 = 3.09×107 copies로 계산된다.
- 대장균 내재유전자 dxs의 경우, 10-2 희석액의 qPCR 평균 PMA-Cq값은 28.48로 계산되었다. 그림1에서 dxs의 표준정량 곡선은 Y = -3.4094x + 43.03로 생산되었다. 이 경우 유전자변형 대장균에 살아있는 균주 세포 수는 10(28.48-43.03)÷-3.4094 × 100 = 1.85×106 copies로 계산된다.
- 따라서 1㎕의 ampicillin 내성 유전자변형 대장균에서 살아있는 유전자변형 대장균 균주 수는 1.85×106이다. 그중 plasmid의 개수는 3.09×107로 계산될 수 있다.
4. 주의사항
- 1) 살아있는 유전자변형 미생물 세포 수의 정량화는 event-specific 유전자(예를 들어, 항생제 내성 유전자)와 taxon-specific 유전자 (예를 들어, bacterial 염색체의 내재유전자)에 대한 표준 정량 곡선을 기반으로 계산되기 때문에 표준 정량 곡선 생산이 매우 중요하다. 모든 매개변수는 ENGL 제안된 표준 범위 내에 있어야 사용할 수 있다.
- 2) Cell-direct qPCR은 DNA 추출 대신 세포 현탁액을 직접 사용하는 qPCR 방법이며, 시료 용액 내의 미생물 분포를 균일하게 유지하는 것이 중요하다. 따라서 모든 배양액 샘플을 vortex 등을 이용하여 균질하게 잘 혼합한 후 취급하여야 한다.
- 3) PMA 용액은 빛에 민감한 차광성 시약이므로 샘플에 첨가할 때 무균작업대의 조명을 끄고 진행한다.
- 4) DNA template 외에 나머지 조성의 시약은 사용하기 작전에 미리 master mix로 만든 다음 필요한 양의 qPCR 반응용액을 PCR 튜브에 옮겨준다. 이는 분석 샘플 간의 실험 오차로 인한 결과에 대한 영향을 최소화한다.
- 5) 무균작업대에서 NTC (Nuclease-free water) PCR well에 시약을 먼저 넣고 밀봉한 다음 나머지 PCR well에 cell 샘플을 첨가하여 실험을 진행한다. 이는 공기 중에서 존재 가능한 미생물의 NTC 오염을 방지하기 위한 예방 조치이다.
- 6) 공기에 부유 세균 등 인한 오염을 방지하기 위하여 배양액 희석, PMA 시약 첨가 및 qPCR 반응액 제작 모두 무균작업대에서 수행한다.
