밀폐이용 안전관리 기술정보
왼쪽메뉴
밀폐이용 안전관리 기술정보
본문컨텐츠
주변토양
유전자변형 미생물 생산시설 주변 토양 모니터링
1. 실험목적
1.1 실험목적
- 유전자변형 미생물 생산공정이용 과정에서 항생제 내성 유전자가 도입된 유전자변형 대장균 (Escherichia coli)과 코리네박테리움 글루타미쿰(Corynebacterium glutamicum), 효모 (Saccharomyces cerevisiae)가 생산 공정 이용시설 주변 토양환경으로 유출될 경우를 가정하여 생산 공정 운영 이후 토양에서 항생제 내성 미생물의 검출 및 밀도 비교 분석 등을 통해 유전자변형 미생물 생산 공정 시에 주변 토양 모니터링 방법을 제시하고자 한다.
1.2 실험 배경
- 유전자변형 미생물 생산공정이용 과정에서 공기 흐름, 작업 인원의 이동 등으로 인해 밀폐공정구역 주변의 외부 토양으로 유전자변형 미생물의 비의도적 유출 가능성이 있다. 기존의 유전자변형 미생물 환경 위해성 평가에서는 PCR 및 NGS 염기서열 분석을 통해 유전자변형 미생물의 존재 여부 또는 수평 전이 여부를 평가하기 위해 토양 DNA를 추출하였으나, DNA 기반기술은 살아있는 유전자변형 미생물과 죽은 유전자변형 미생물을 구분할 수 없는 단점이 있다. 따라서, 살아있는 생산 균주의 밀폐공정에서의 비의도적 유출 여부의 관리를 위해서는 살아있는 유전자변형 미생물을 직접 모니터링 하는 것이 아주 중요하다. 이를 위하여, 국내에서 유전자변형 생산 균주의 주요 숙주로 이용되는 미생물인 그람 음성균인 대장균, 그람 양성균인 코리네박테리움 글루타미쿰, 진균류 효모를 숙주로 한 유전자변형 미생물의 토양 중의 비의도적 유출을 가정하고, 먼저 항생제 마커에 대한 항생제 선택배지를 이용하여 살아있는 항생제 내성 미생물을 선택적으로 선별하였다. 이어서 유전자변형 미생물의 event-specific 유전자 염기서열 및 taxon-specific 유전자 염기서열을 기반으로 설계된 oligo primer를 이용하여 일차적으로 선별된 항생제 내성 콜로니들의 cell-direct PCR 분석을 통해 DNA의 추출 없이 살아있는 유전자변형 미생물의 존재를 확인하는 실용적인 방법을 제시하였다.
2. 실험장비(도구) 및 재료
2.1 실험기자재
- - 토양 오거(Soil auger, 토양시료 채취기): 길이 1m, 직경 50mm
- - 전자저울
- - 냉장고
- - 항온배양기
- - 볼텍스믹서
- - Thermal Cycler
- - Micro pipette (1-10㎕, 20-200㎕, 100㎕-1000㎕, 1000㎕-5000㎕)
- - 소형 전기영동장치(Mini Submarine Electrophoresis System)
- - 고압증기멸균기
- - 무균작업대
- - 진탕 배양기
- - PCR tube (0.2㎖)용 Micro centrifuge
- - Ultraviolet (UV) Transilluminator
2.2 실험재료
- - 토양 채취용 지퍼백
- - 50 ㎖ Conical 튜브
- - PCR tube (200㎕)
- - Micro pipette tip (1-10㎕, 20-200㎕, 100㎕-1000㎕, 1000㎕-5000㎕)
- - Tryptic Soy Agar (TSA)
- - Luria-Bertani (LB) agar
- - Brain Heart Infusion (BHI)
- - Yeast Extract Peptone Dextrose (YPD) agar
- - 유전자변형 (GM) 미생물의 선발표지유전자 해당 항생제, 예를 들어 50ug/㎖ Kanamycin (E.coli and C.glutamicum), 200ug/㎖ G418 (S.cerevisiae)
- - 항진균제 50ug/㎖ cycloheximide
- - 미생물 배양용 멸균 Petri dish
- - 1.5㎖ Tube
- - 멸균증류수 (SDW, sterilization distilled water)
- - Oligo primer
- - 1.5% agarose gel
- - SafePinky DNA gel staining solution (10000×) in water
- - SapphireAmp ® Fast PCR Master Mix (2×Premix) (Bacterial E.coli, C.glutamicum)
- - Phire TM Plant Direct PCR Master Mix (진균류 효모)
- - Spreader
- - 100 bp DNA Ladder
- 참고사항:
- 1. 위에서 제시한 SapphireAmp® Fast PCR Master Mix (2×Premix) kit를 이용하여 bacterial 세포 direct PCR이 가능하며, 일반적으로 상용화된 Taq DNA 중합효소를 이용하여 PCR도 가능하다.
- 2. 진균류 효모 세포 direct PCR의 경우, 일반적인 Taq DNA polymerase는 효모 세포 잔해물과 배양액의 성분에 의한 PCR 억제로 PCR 증폭이 어렵다. 이러한 이유로 앞서 제시한 PhireTM Plant Direct PCR Master Mix에서는 Phire™ Hot Start II DNA 중합효소가 많은 억제제 내성을 가지고 있어 진균류 세포의 direct PCR에 이용할 것을 권장한다.
3. 실험과정
3.1 유전자변형 미생물 공정 주변 토양 채취
- 1) 토양 시료는 해당 유전자변형 미생물을 대량으로 배양하는 밀폐생산 시설 주변의 지역 중 누출될 가능성이 있는 곳을 선정한다. 통상적으로 생산시설의 주 출입구 부근의 토양은 작업자들의 빈번한 출입으로 인해 유전자변형 미생물의 비의도적 유출 가능성이 높음으로 주 출입구 양측에서 토양을 채취하는 것을 권장한다. 만약 유전자변형 미생물이 발견될 경우 더 넓은 범위로 토양 모니터링을 확대한다.
- 2) 유전자변형 미생물의 밀폐시설에서의 유출 여부의 확인을 위해서는 공정 전 토양 시료를 대조군으로 공정 후 토양 시료를 시험군으로 하여 분석하는 것을 권장한다.
- 3) 토양 시료 채취 위치는 주 출입구를 기준으로 좌우 양측 30cm 떨어진 거리에서 1M 간격으로 3개 지점을 선정하고, 공정운영 후에는 공정 전 샘플 채취 지점 바로 옆의 토양에서 채취한다(그림 1).
- 4) 선정된 지점에서 soil auger 등 토양 채집기(그림 2)를 이용하여 10cm 깊이, 직경 50mm의 낙엽층과 유기물층을 포함하는 토양 시료를 채집한 후 지퍼백에 밀봉하여 실험실로 운반 후 4℃에 보관한다.
- 5) 실험실로 운반한 토양 시료 1g을 50㎖ 튜브에 담고, 멸균증류수 18㎖를 첨가하여 10-1로 희석한 후 뭉친 토양 시료가 없도록 볼텍스믹서를 이용하여 교반한다.
- 6) 토양 시료는 25℃의 진탕 배양기에서 30분간 진탕 배양한다.
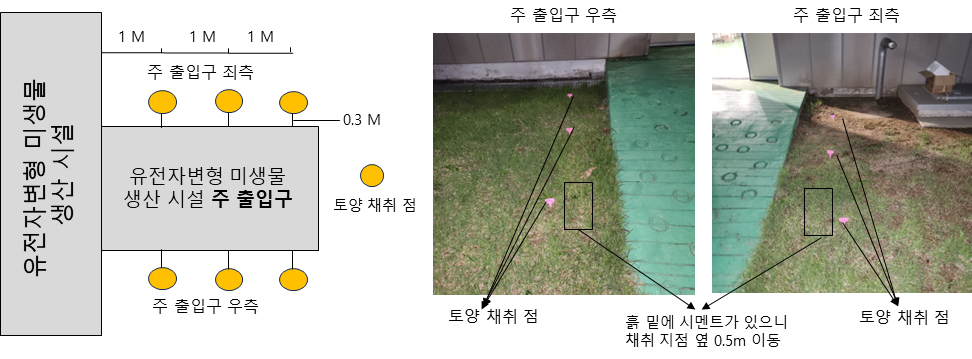
- 그림 1. 유전자변형 미생물 생산시설 주변 토양 모니터링 채집 위치 예시도

- 그림 2. Soil auger
3.2 토양에 항생제 내성 미생물 밀도 분석
- 1) 무균작업대에서 토양 시료를 10-4까지 희석한다. 10-2 희석액은 1㎖의 10-1 토양 희석액에 9㎖ 멸균증류수를 첨가하여 30초간 볼텍스믹서를 이용하여 교반하여 제작한다. 동일한 방식으로 10-4 까지의 희석액을 준비한다(그림 3).
- 2) 유전자변형 미생물의 선발표지 유전자 해당 항생제(kanamycin 등)를 첨가한 각 미생물별 최적 고체배지에 10-2 에서 10-4까지 각각의 토양 희석액 100㎕를 3 반복으로 도말한다. 도말 전 토양 희석액은 30초간 교반하여 사용한다.
- 3) 토양 희석액이 도말된 고체 배지는 30℃ 항온배양기에서 48시간 동안 배양한다.
- 4) 저항성 코로니 계수가 적절한 희석 배지를 선정하여 아래의 공식을 기반으로 3회 반복의 평균 콜로니 수를 측정하여 코로니 집락형성능 (Colony Forming Unit, CFU)를 계산한다.

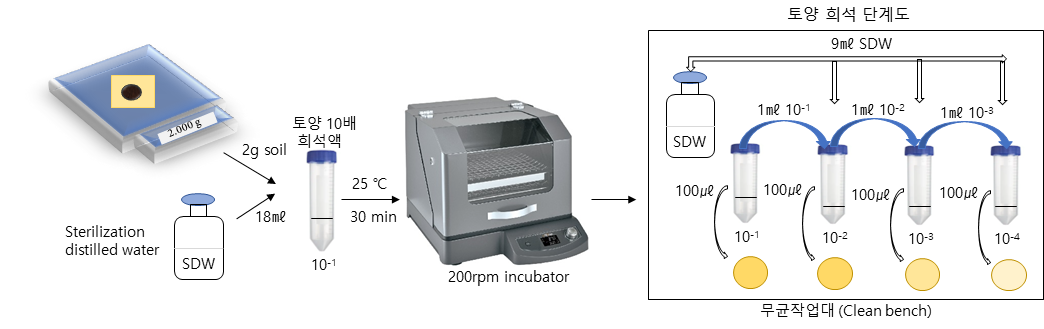
- 그림 3. 토양 시료의 희석 단계 예시도.
3.3 토양 중의 항생제 내성 미생물 수집 및 정성 PCR 분석
- 1) 토양 내에는 자연계에 존재하는 다양한 항생제 저항성 균이 존재하기 때문에 항생제 저항성을 나타내는 코로니가 모두 유전자변형 미생물은 아니다. 토양 중 항생제 내성 미생물에 유전자변형 미생물이 존재하는지 확인하기 위해서는 토양 채취 지점들의 10-2 희석액을 도말한 미생물 배지 1개에 멸균증류수 1㎖를 넣고 spreader 이용해서 배지 위에 있는 미생물을 현탁한다.
- 2) 현탁액을 1.5㎖ 튜브에 옮긴 뒤 1분간 교반하여 PCR 분석을 수행한다.
- 3) PCR 분석은 각 유전자변형 미생물의 도입유전자 event-specific 염기서열과 taxon-specific 유전자(내재 유전자)를 기반으로 제작된 프라이머를 사용한다. 대장균, 코리네박테리움, 효모의 단일 복사본 내재유전자 예시는 표 1과 같다.

- 표 1. 유전자 변형 미생물에 대한 내재 유전자 검출용 primer 서열 정보 (예시)
- 4) 대장균과 코리네박테리움 PCR 반응액 조성은 표 2와 같다.
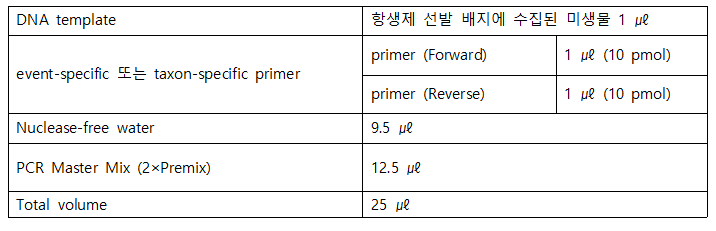
- 표 2. 유전자 변형 대장균 및 코리네박테리움 PCR 반응 조성표 예시
- 5) 진균류 효모 (S. cerevisiae)의 경우 PhireTM Plant Direct PCR Master Mix (Thermo ScientificTM)를 사용하여 PCR 분석을 권장한다. PCR 반응액 조성은 표 3과 같다.
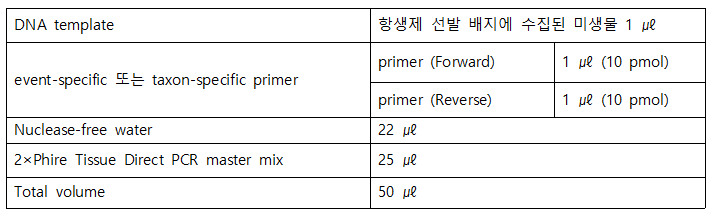
- 표 3. 유전자변형 효모 PCR 반응 조성표 예시
- 6) PCR 반응물의 전기영동 후 event-specific과 taxon-specific에서 모두 PCR 증폭이 발생한 경우 발생량 검정을 위해 각각의 코로니에 대한 PCR 분석이 요구된다.
- 7) 희석 배양된 배지 중 단독 코로니 분리가 가능한 배지를 선택하여 10개의 코로니를 선택하여 표2와 표3의 조성표에 준하여 colony PCR를 수행한다.
- 8) PCR로 분석된 유전자변형 미생물의 수를 측정하고 희석 배수, 전체 코로니 중 양성 코로니 수 등을 적용하여 비율을 계산한다.
4. 주의사항
- 1) 선정한 토양 채취 지점의 토양에서 돌 등 이물질이 있을 경우 이를 피하여 해당 지점 근처로 이동하여 토양 시료를 채취한다. 단, 공정 전후 동일한 지점에서 토양 채취한다.
- 2) Event-specific PCR 프라이머는 도입유전자와 주변 염기서열 부위를 포함하게 디자인해야 한다.
